Makina Blog
Calculez sur GPU avec Python – Partie 3/3
L’architecture d’un GPU
Même si certaines librairies comme Cupy, Numba ou PyOpenCL permettent de lancer des calculs pour GPU de manière « simplifiée », il est important de comprendre l’architecture d’un processeur graphique pour organiser les calculs qu’il devra accomplir.
À défaut les performances ne seront pas optimales, et, même pire, le temps de calcul pourrait être plus lent que sur CPU, comme nous venons de l’observer.
La mémoire
Nous l’avons déjà évoqué, mais il bon de le rappeler et préciser :
- Un processeur graphique calcule avec sa propre mémoire interne
- Il n’utilise pas la mémoire du CPU
Votre GPU doit donc disposer d’une mémoire suffisante pour calculer avec de grandes matrices/images/vecteurs, enfin, avec des données volumineuses. Sinon vous obtiendrez des Memory Overflow et ne pourrez mener à terme vos calculs.
Les échanges de données entre le CPU et le GPU sont lents. Il convient de les minimiser au maximum, sinon ils viendront diminuer drastiquement vos performances.
Nous avons déjà vu comment obtenir la mémoire dont dispose le GPU :
print( "%s Mega Bytes" % (gpu0.total_memory() // 1024**2))
7766 Mega Bytes
Nous pouvons reprendre la petit calcul du produit des vecteurs a et b en réalisant réalisant nous-mêmes le transfert mémoire du CPU vers le GPU afin de mieux mesurer ces temps.
Dans le calcul réalisé avec la fonction multiply_them le temps de transfert des données CPU vers GPU puis GPU vers CPU était inclus dans la mesure. Ce n’était pas le cas avec cupy car les vecteurs ac, bc et destc ont été alloués directement dans le GPU.
Le code cupy était donc plus rapide, et ceci d’un facter x16 par rapport à la fonction native. C’est dire si ce temps de transfert des données est coûteux.
Vérifions cela avec multiply_them en créant les vecteurs dans le GPU avant d’appeler la fonction.
La fonction mem_alloc permet d’allouer de la mémoire sur le GPU.
a_gpu = drv.mem_alloc(a.nbytes) # Allocating a.nbytes bytes for a gpu array named a_gpu
b_gpu = drv.mem_alloc(b.nbytes) # Same for b/b_gpu
Nous disposons maintenant de 2 tableaux a_gpu et b_gpu alloués dans la ram du GPU.
Il convient maintenant de leur transférer les données des vecteurs Numpy a et b
La fonction memcpy_htod transfère les données du CPU vers le GPU.
hsignifie ici : Host, il s’agit du CPUdsignifie ici : Device, il s’agit du GPU
Host et Device sont des termes de la terminologie CUDA pour désigner le CPU qui lance les calculs et le GPU qui est le périphérique (device) qui les exécute.
Nous y reviendrons tout à l’heure.
drv.memcpy_htod(a_gpu, a) # Copying data from numpy array in RAM to GPU RAM
drv.memcpy_htod(b_gpu, b) # Same for b
dest_gpu = drv.mem_alloc(a.nbytes) # Also need to allocate memory for the result
Nous avons aussi alloué la mémoire pour le tableau résultat dest_gpu.
Par contre aucune donnée n’y a été copiée puisqu’il est utilisé pour recevoir le résultat.
Relançons le calcul.
multiply_them( dest_gpu, a_gpu, b_gpu, block=(400, 1, 1), grid=(1, 1) )
Vérifions le résultat en transférant avec la fonction memcpy_dtoh les données du device vers l’hôte.
dest[:] = 0
print(dest[:5])
drv.memcpy_dtoh(dest, dest_gpu)
print(dest[:5])
[0. 0. 0. 0. 0.]
[ 0.06958307 -0.01390467 1.4210043 0.84489673 0.4126398 ]
print(sum(a*b - dest)) # Ok, still good !
0.0
Mesurons le temps d’exécution…
%timeit multiply_them( dest_gpu, a_gpu, b_gpu, block=(400, 1, 1), grid=(1, 1) )
6.22 μs ± 59.4 ns per loop (mean ± std. dev. of 7 runs, 100,000 loops each)
Nous avons gagné et dépassé les performances affichées par CuPy. Ce qui est normal, la classe vecteur de CuPy ajoute un peu plus de code pour gérer ses structures de données alors que la fonction multiply_them est la plus minimaliste possible.
Cela signifie aussi que le temps de transfert des données représentait environ 95% du temps de calcul : c’est un temps très coûteux qu’il convient de minimiser au mieux.
Mais nous restons 14 fois plus lent que le CPU !
6.22/0.422
14.739336492890995
La librairie PyCuda dispose d’une classe gpuarray qui permet de manipuler des tableaux CUDA à la manière de NumPy.
Elle facilite aussi grandement les échanges de mémoire entre CPU et GPU.
Elle implémente aussi la majorité des opérateurs NumPy, comme +, -, /, * ou encore **.
Une fois les vecteurs créés, vous les utilisez comme un tableau NumPy.
from pycuda import gpuarray
gpu_a = gpuarray.to_gpu(a)
gpu_b = gpuarray.to_gpu(b)
gpu_dest = gpu_a * gpu_b
cpu_dest = gpu_dest.get() # transfert GPU array to CPU array of numpy type
gpu_dest[:5] # Convert gpu_dest to numpy array too
array([ 0.06958307, -0.01390467, 1.4210043 , 0.84489673, 0.4126398 ],
dtype=float32)
%timeit gpu_dest = gpu_a * gpu_b
34.6 μs ± 2.71 μs per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
Évidemment cela ne change rien aux mesures observées jusqu’ici…
Clairement, il nous manque encore des éléments pour comprendre comment tirer profit de ces GPU…
En fait, il y a plusieurs éléments qui entrent en jeu pour expliquer ces mesures, dont 2 majeurs ici:
- Le code utilisé sur le GPU est généralement compilé par PyCuda au premier appel ce qui vient ternir les performances la première fois.
L’utilisation de%prunle montrera si vous faites l’expérience dans une nouvelle session.
Puis les fois suivantes le code compilé est réutilisé depuis le cache, ce qui améliore nettement les mesures faites sur le GPU.
Dans notre cas nous utilisons%timeitqui appelle de très nombreuses fois la fonction, donc le temps de compilation n’est plus visible car il est dilué sur l’ensemble des mesures.
Il n’explique donc pas ces moindres performances du GPU. - La taille des données: ici les données sont de toutes petites taille, elles peuvent résider dans la mémoire cache du CPU ce qui compense largement le manque de cœurs sachant que la fréquence du CPU est 3 fois supérieure à celle du GPU et que le CPU dispose d’instructions SIMD.
Recommençons l’exercice en faisant varier le nombre d’éléments des tableaux a et b et de leurs équivalents gpu_a et gpu_b de 1 à 100_000_000 d’éléments.
Nous allons, pour chaque tableau a/b et gpu_a/gpu_b: * mesurer le temps de calcul de leur produit * calculer les ratios temps CPU/GPU et inversement * tracer ces durées sur un graphique en fonction du nombre d’éléments
Remarque
Pour utiliser des tableaux d’un milliard d’éléments il faudrait disposer de 12 Go de RAM dans le GPU car nous avons 3 tableaux de réels 32 bits (4 octets) qui doivent tenir dans la RAM de la carte graphique. Ce n’est pas le cas avec la carte utilisée lors de la rédaction de ce TP.
Dans le code ci-après, nous réalisons 10 mesures pour chaque tableau afin d’avoir un temps plus précis et conservons la plus petite valeur pour obtenir les meilleurs ratios.
%matplotlib inline
import time
import numpy as np
import matplotlib.pyplot as plt
from ipywidgets import interact
from pycuda import gpuarray
from ipywidgets import interactive
cpu_time = []
gpu_time = []
nb_items = []
for n in range(9):
a = np.random.random(10**n).astype(np.float32)
b = np.random.random(10**n).astype(np.float32)
nb_items.append(n)
# move data to gpu memory
gpu_a = gpuarray.to_gpu(a)
gpu_b = gpuarray.to_gpu(b)
gpu_d = gpuarray.empty_like(gpu_a)
cpu = []
gpu = []
# Do 10 timings and get min time
for repeat in range(10):
start = time.time()
dest = a * b
stop = time.time()
duration = stop - start
cpu.append(duration)
start = time.time()
gpu_d = gpu_a * gpu_b
stop = time.time()
duration = stop - start
gpu.append(duration)
cpu_time.append(min(cpu))
gpu_time.append(min(gpu))
# Displaying
fig, ax1 = plt.subplots(figsize=(12,4))
ax1.plot(nb_items, cpu_time, label="CPU time to compute vector product", c='blue')
ax1.plot(nb_items, gpu_time, label="GPU time to compute vector product", c='red')
ax1.set_ylabel("Compute time in seconds")
ax1.legend()
ax2 = ax1.twinx()
ax2.set_ylabel("Ratio CPU vs GPU")
ax2.plot(nb_items, np.array(cpu_time) / np.array(gpu_time), label="Ratio CPU/GPU", c='orange')
ax2.plot(nb_items, np.array(gpu_time) / np.array(cpu_time), label="Ratio GPU/CPU", c='cyan')
ax2.legend(loc='center')
ax2.tick_params('y', colors='black')
fig.tight_layout()
ax1.set_xlabel('log10(vector shape)')
plt.show()
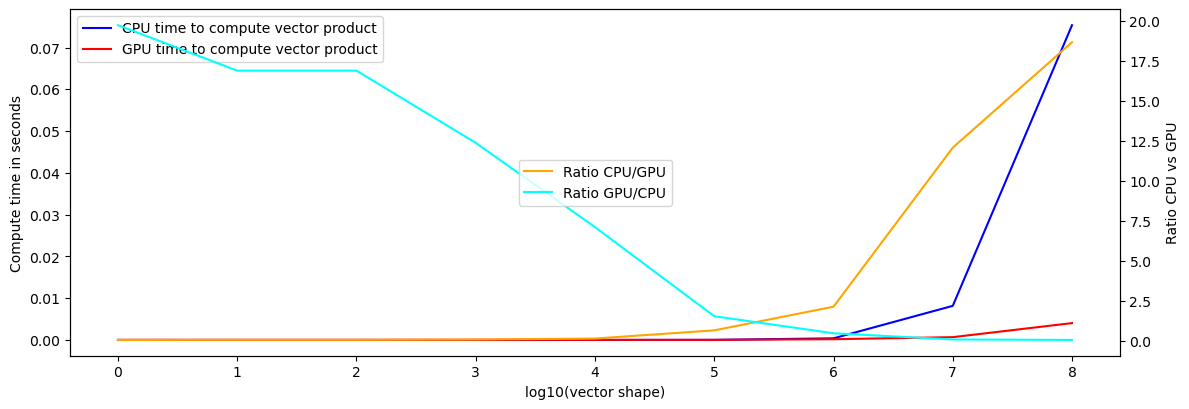
Il apparaît clairement que sur les tableaux de moins d’un million d’éléments l’avantage est nettement en faveur du CPU. Mais dès que l’on dépasse ce million, le temps de calcul du CPU monte en flèche alors que pour le GPU il reste presque contant du début à la fin.
Et nous finissons par avoir un ratio d’un peu plus de 17.5 fois plus rapide en faveur du GPU ! C’est notre ratio théorique estimé précédemment !
Trop fort ! Quand la théorie est confirmée par la pratique, c’est toujours un grand moment de science !
Question: Pourquoi le rapport de performance s’inverse vers 1 million d’éléments en faveur du GPU ?
Cela est lié à 2 choses:
- La mémoire cache du CPU
- La grande taille du tableau qui permet d’utiliser tout le potentiel des milliers de cœurs du GPU
La mémoire de votre ordinateur est stockée dans des barrettes mémoire sur la carte mère. Elle ne réside pas dans le CPU comme nous l’avons vu dans le chapitre sur les concepts du parallélisme.
Cette mémoire est généralement cadencée à une fréquence inférieure à celle du CPU: Imaginons une RAM cadencée à 1Ghz contre 3Ghz pour le CPU.
Cela signifie qu’en une seconde votre CPU peut effectuer 3 fois plus de cycles que votre RAM. Et c’est problématique.
- Quand le CPU veut incrémenter le contenu d’un octet situé en RAM, il va charger dans un de ses registres son contenu pour le modifier, car la RAM ne permet que de stocker des valeurs, pas de calculer directement dessus.
Certaines RAM disposent de telles opérations, mais elles sont plus coûteuses.- Puis le CPU incrémente, la valeur stockée dans son registre
- Et il la redépose dans la RAM
La durée de ces opérations sera la suivante: – 1 cycle RAM pour le transfert de l’octet vers le CPU. Cela représente 3 cycles ou le CPU ne fait rien et attend – 1 cycle de calcul pour le CPU – 1 cycle RAM pour le transfert du registre dans la RAM. Cela représente 3 cycles ou le CPU ne fait rien et attend
Au total cette opération aura requis 7 cycles du CPU sachant que pendant 6 d’entre eux il attend la fin des opérations dans la RAM.
Donc, si la RAM avait fonctionné à la même fréquence que le CPU l’opération n’aurait duré pour le CPU que 3 cycles, contre 7. Aussi, votre CPU va tourner plus de 2 fois moins vite que ce qu’il pourrait faire en raison de la lenteur de la mémoire externe. Et votre ressenti sera une fréquence de 1,5Ghz ! Ce ne sera pas un défaut du CPU mais une trop grande lenteur de votre RAM.
Pour éviter ces attentes inutiles liées au fait que la RAM dispose d’une fréquence inférieure, les CPU modernes possèdent une mémoire interne, dite mémoire cache de type SRAM qui est bien plus rapide et permet au CPU de tourner à sa fréquence optimale. Mais elle est bien plus petite : quelques Mo au mieux car elle est stockée directement dans le CPU et qu’il y a peu de place dans la puce – et puis elle est bien plus chère.
Donc, tant que le CPU peut faire tourner les données qu’il manipule dans sa mémoire cache, il tourne à pleine fréquence et ses performances sont optimales.
Dès que les données à manipuler deviennent plus volumineuses, il fait de nombreux aller-retour avec la mémoire externe et ses performances se dégradent.
Si l’on compte 3 tableaux de 1 million de réels (a, b et dest) en simple précision (4 octets), cela occupe très exactement 12 000 000 d’octets.
Observons la mémoire du CPU avec la commande lscpu ou la documentation officielle du processeur utilisé :
!lscpu| grep -i cache
Cache L1d : 192 KiB (6 instances)
Cache L1i : 192 KiB (6 instances)
Cache L2 : 1,5 MiB (6 instances)
Cache L3 : 12 MiB (1 instance)
Vulnerability L1tf: Mitigation; PTE Inversion; VMX conditional cache flushes, SMT vulnerable
Chaque cœur dispose de caches individuels de 192Kox2 puis 1,5Mo et enfin peuvent partager 12Mo supplémentaires.
Nos 3 tableaux d’un million d’éléments peuvent parfaitement tenir dans le cache du processeur, au-delà il va chercher les données en RAM et ses performances vont se dégrader alors que le GPU va pouvoir déployer tous ses cœurs sur ses millions de données et tirer pleine partie de son architecture ultra parallélisée !
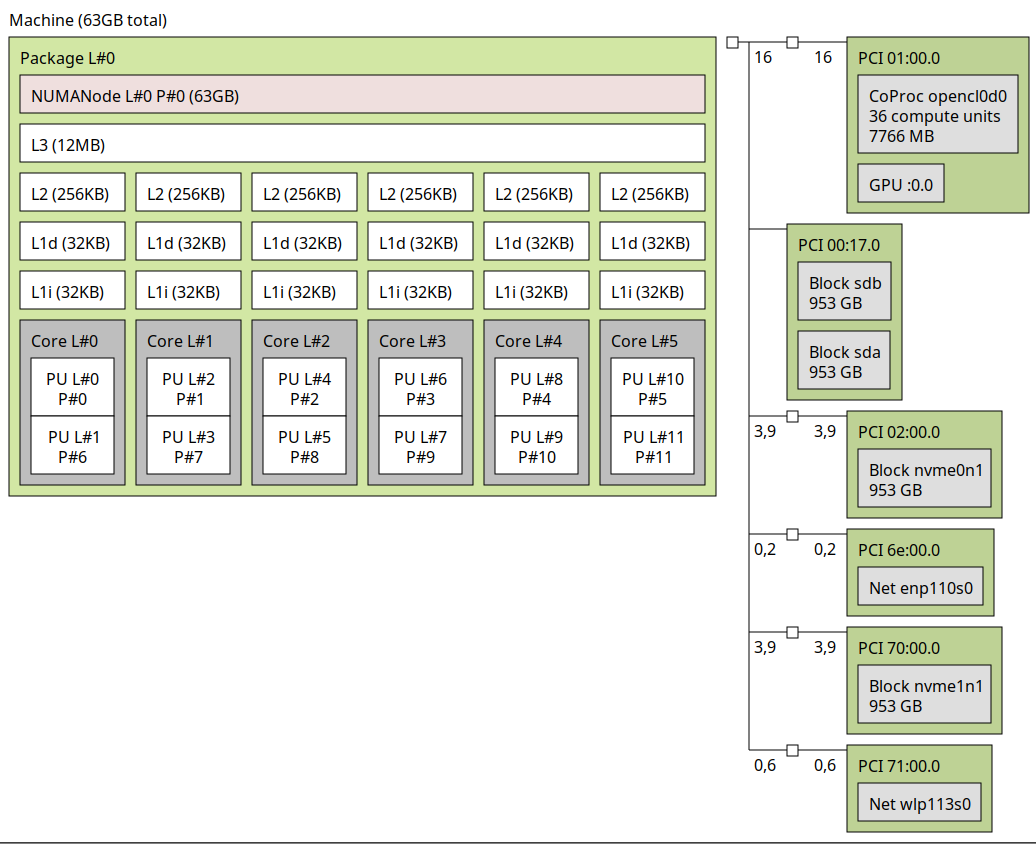
La commande hwloc-ls vous permet d’avoir une vision plus fine de la composition de votre processeur.
$ hwloc-ls --of console
Machine (31GB)
Package L#0 + L3 L#0 (12MB)
L2 L#0 (256KB) + L1d L#0 (32KB) + L1i L#0 (32KB) + Core L#0
PU L#0 (P#0)
PU L#1 (P#6)
L2 L#1 (256KB) + L1d L#1 (32KB) + L1i L#1 (32KB) + Core L#1
PU L#2 (P#1)
PU L#3 (P#7)
L2 L#2 (256KB) + L1d L#2 (32KB) + L1i L#2 (32KB) + Core L#2
PU L#4 (P#2)
PU L#5 (P#8)
L2 L#3 (256KB) + L1d L#3 (32KB) + L1i L#3 (32KB) + Core L#3
PU L#6 (P#3)
PU L#7 (P#9)
L2 L#4 (256KB) + L1d L#4 (32KB) + L1i L#4 (32KB) + Core L#4
PU L#8 (P#4)
PU L#9 (P#10)
L2 L#5 (256KB) + L1d L#5 (32KB) + L1i L#5 (32KB) + Core L#5
PU L#10 (P#5)
PU L#11 (P#11)
Elle propose même un aperçu graphique.
Fonctions ElementwiseKernel
Nous avons utilisé la classe gpu_array qui facilite l’usage des tableaux pour le GPU, mais en utilisant les fonctions dites ElementwiseKernel fournies par CUDA nous pouvons encore améliorer les choses.
Ces fonctions sont des fonctions C, très courtes, très rapides conçues pour ne travailler que sur un seul élément, indépendamment des autres, ce qui permet des optimisations très avancées du compilateur.
from pycuda.elementwise import ElementwiseKernel
gpu_mul_ker = ElementwiseKernel(
"float *in1, float *in2, float *out",
"out[i] = in1[i]*in2[i];",
"gpu_mul_ker"
)
gpu_mul_ker est une fonction C :
- Elle prend 3 tableaux de réels simple précision en paramètres
- Les 2 premiers sont les tableaux à multiplier
- Le dernier contiendra le résultat
- Le code tient sur une ligne, le paramètre
iest géré par CUDA, il s’agit du cœur qui fera le calcul
La fonction retournée peut être utilisée avec des tableaux situés dans la RAM du GPU :
a = np.random.random(10**6).astype(np.float32)
b = np.random.random(10**6).astype(np.float32)
# move data to gpu memory
gpu_a = gpuarray.to_gpu(a)
gpu_b = gpuarray.to_gpu(b)
gpu_d = gpuarray.empty_like(gpu_a)
# vérifions le résultat
gpu_mul_ker(gpu_a, gpu_b, gpu_d)
gpu_d - gpu_a*gpu_b
array([0., 0., 0., ..., 0., 0., 0.], dtype=float32)
%matplotlib inline
import time
import numpy as np
import matplotlib.pyplot as plt
from ipywidgets import interact
from pycuda import gpuarray
# from ipywidgets import interactive
cpu_time = []
gpu_time = []
nb_items = []
for n in range(9):
a = np.random.random(10**n).astype(np.float32)
b = np.random.random(10**n).astype(np.float32)
nb_items.append(n)
# move data to gpu memory
gpu_a = gpuarray.to_gpu(a)
gpu_b = gpuarray.to_gpu(b)
gpu_d = gpuarray.empty_like(gpu_a)
cpu = []
gpu = []
# Do 10 timings and get min time
for repeat in range(10):
start = time.time()
dest = a * b
stop = time.time()
duration = stop - start
cpu.append(duration)
start = time.time()
gpu_mul_ker(gpu_a, gpu_b, gpu_d)
stop = time.time()
duration = stop - start
gpu.append(duration)
cpu_time.append(min(cpu))
gpu_time.append(min(gpu))
# Displaying
fig, ax1 = plt.subplots(figsize=(12,4))
ax1.plot(nb_items, cpu_time, label="CPU time to compute vector product", c='blue')
ax1.plot(nb_items, gpu_time, label="GPU time to compute vector product", c='red')
ax1.set_ylabel("Compute time in seconds")
ax1.legend()
ax2 = ax1.twinx()
ax2.set_ylabel("Ratio CPU vs GPU")
ax2.plot(nb_items, np.array(cpu_time) / np.array(gpu_time), label="Ratio CPU/GPU", c='orange')
ax2.plot(nb_items, np.array(gpu_time) / np.array(cpu_time), label="Ratio GPU/CPU", c='cyan')
ax2.legend(loc='center')
ax2.tick_params('y', colors='black')
fig.tight_layout()
ax1.set_xlabel('log10(vector shape)')
plt.show()
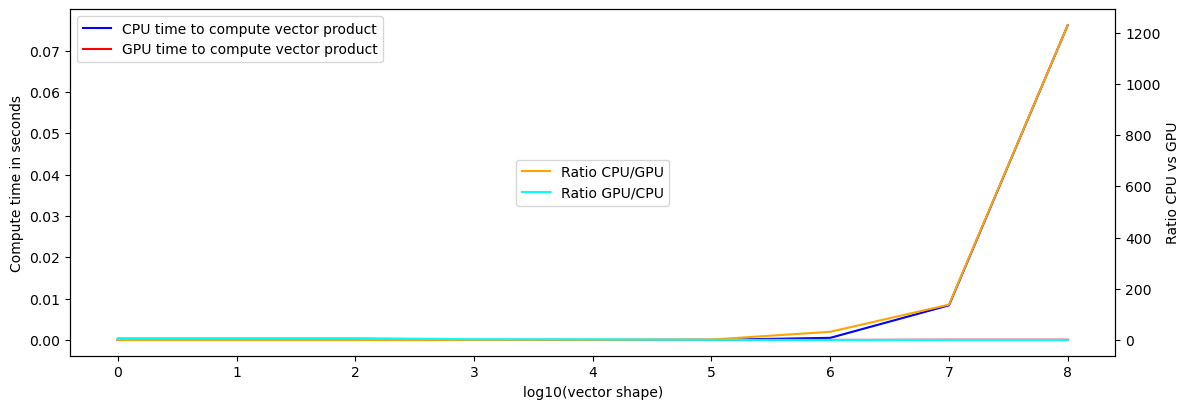
Cette fois, nous mesurons la toute puissance de la carte graphique qui est plus de 1_000 fois plus rapide que le CPU.
Sur les tableaux de moins d’un million d’éléments le CPU a toujours le dessus. Ce n’est plus visible en raison de l’échelle mais si vous diminuez la valeur de N, vous verrez que cela n’a pas changé.
Par contre dès que nous dépassons le million d’éléments, le ratio explose en faveur du GPU, qui est environ 1200 fois plus rapide que le CPU !
Utilisez les GPU avec de très grandes volumétries de données
Pour vraiment bénéficier de l’architecture massivement parallèle de ces processeurs il faut pouvoir effectuer des calculs très nombreux en parallèle, sinon ils perdent tout leur intérêt.
C’est la conclusion à retenir !
En dessous d’un million d’éléments/calculs parallélisables un GPU ne sera probablement pas plus intéressant qu’un CPU.
Noyaux (Kernels), Threads, Blocks, et Grilles (Grids)
L’architecture matérielle d’un GPU est différente d’un CPU.
CPU
Nous savons qu’un CPU possède des cœurs, ou unités de calcul autonomes, qui peuvent se décomposer en threads, en général 2. Le CPU possède une mémoire cache décomposée en niveau de quelques Ko ou Mo. Une partie de cette mémoire cache est propre à chaque cœur, tandis que le dernier niveau est partagé par tous les cœurs. Cette mémoire est chargée avec les données de la RAM via des bus de données PCI. Ce qui est une source de ralentissement du CPU.
- Chaque cœur du CPU est autonome
- Dans un même cœur un seul thread est actif à la fois, et quand il est en attente de données (mémoire, disque, réseau, …), l’autre thread prend la main.
GPU
Sur les cartes NVIDIA, l’architecture logique est basée sur la notion de Kernel, Thread, Blocks et Grids.
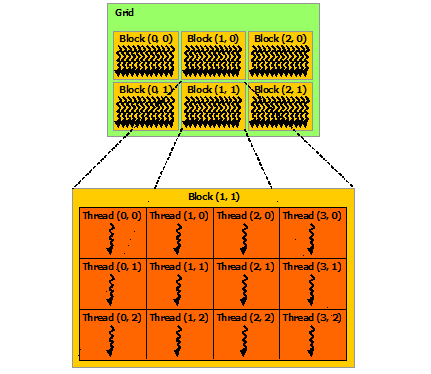
Source* Wikipédia/NVIDIA Threads, Blocs et Grilles
Nous les nommerons parfois avec la terminologie française : noyau, fil d’exécution/fonction, Bloc et Grille.
- Un Kernel ou noyau CUDA est une fonction parallélisée qui peut être lancée directement depuis le processeur hôte (le CPU) sur le device (le GPU).
Elle a pour but de lancer les calculs sur l’ensemble de la grille via des fonctions device. - Une fonction device est une fonction appelée par la fonction du Kernel (ou une autre fonction device).
Ces fonctions peuvent être comparées aux fonctions classiques d’un programme C, excepté qu’elles sont exécutées sur le GPU et initiées par une fonction kernel (exécutée par le CPU). - Un Thread est une fonction qui s’exécute sur un des cœurs du GPU.
Un thread exécute donc une fonction device. - Un Bloc/Block est un groupe de threads : dans la philosophie ils sont semblables aux chunks de dask si vous connaissez cette librairie
- Une Grille/Grid est un groupe de blocks : dans la philosophie la grille est semblable au tableau dask composé de chunks
Votre carte graphique dispose de milliers de cœurs. 2304 pour la carte RTX2070 utilisée dans notre exemple.
Si vous devez incrémenter de 1 chaque valeur d’un tableau disposant d’un million d’éléments, vous n’avez pas assez de cœurs pour chacune des cases.
L’idée est donc de découper ce tableau (la grille) en blocs : * Chaque bloc possède un nombre de cœurs et travaillera sur une portion du tableau * Et les cœurs passeront de bloc en bloc pour traiter l’ensemble de la grille
Par exemple, si vous disposez d’une carte avec 1000 cœurs, vous pourriez faire des blocs de 100 cœurs chacun. Avec 10 blocs vous utiliseriez tous vos cœurs.
Votre tableau serait alors composé de 10_000 blocs et comme vous avez 1000 cœurs, vous pourriez lancer vos calculs en parallèle par paquets de 10 blocs à la fois et répéter ainsi 1000 fois l’opération.
C’est ainsi que fonctionnent les cartes graphiques.
Mais que de questions…
- Comment choisir le découpage de ma grille en blocs et de mes blocs en Threads ?
- Il y a-t-il des limites pour le nombre de blocs dans une grille, de Threads/cœurs dans un bloc ?
- Combien de blocs peut-on lancer simultanément en parallèle ?
Ce sont de bonnes questions car ces éléments dépendent des spécifications de votre carte graphique.
Vous trouverez les chiffres relatifs à votre carte dans le tableau compute capablity de la documentation CUDA.Mais les questions les plus redoutables sont peut-être celles-là : * Comment utiliser au mieux mes X cœurs si le nombre d’éléments de mon tableau n’est pas multiple de X, disons qu’il vaut X+1 ,.5X ou 2X-1 ? * Comment découper ma grille dans ce cas ?
La réponse suit dans l’exemple ci-après.
Streaming Multiprocessors (SM)
Un Streaming Multiprocessor représente un ensemble de cœurs qui pourront traiter un même flux de données. C’est un peu le nombre de blocs que vous pourriez lancer simultanément.
L’attribut MULTIPROCESSOR_COUNT de la carte graphique indique le nombre de SM dont elle dispose :
gpu0 = drv.Device(0)
gpu0.get_attribute(drv.device_attribute.MULTIPROCESSOR_COUNT) # String 'MULTIPROCESSOR_COUNT' is not allowed
36
La carte peut donc traiter 36 flux en parallèle.
Le nombre de cœurs de chaque SM est défini par les capacités de calcul de votre carte graphique.
Ces capacités sont définies par un numéro de version qui précise les fonctionnalités supportées par le matériel.
gpu0.compute_capability()
(7, 5)
print(gpu0.get_attribute(drv.device_attribute.COMPUTE_CAPABILITY_MAJOR))
print(gpu0.get_attribute(drv.device_attribute.COMPUTE_CAPABILITY_MINOR))
7
5
La version des capacités de cette carte graphique est donc 7.5.
Pour connaître les fonctionnalités associées il faut se référer à la documentation CUDA.
La capacité de calcul 7.5, qui correspond à l’architecture Turing, permet d’utiliser 64 cœurs par SM pour des calculs sur des réels en simple précision et 32 cœurs par SM pour des calculs sur des réels en double précision.
Soit un total de 2304 CUDA cores pour calculer sur des réels simple précision.
C’est bien ce que précise la page de présentation de la carte graphique RTX 2070
print(36*64) # We find the 2304 cuda cores displayed on NVIDIA RTX 2070 web page
2304
Threads
Un Thread est une suite d’instructions qui sont exécutées sur un seul cœur. Un thread n’est pas un cœur. C’est une fonction. Le GPU peut recevoir plus de threads qu’il ne possède de cœurs, tout comme un CPU exécute plus de programmes qu’il n’a de cœurs.
Blocks
Les Threads sont exécutés dans des unités abstraites appelées blocs. À" l’intérieur d’un bloc leTthread peut être indexé par une deux ou trois dimensions : threadIdx .x, .y et .z
Un block ne peut pas contenir plus de 1024 Threads.
Grids
Les blocs sont quant-à eux exécutés dans d’autres unités abstraites appelées grilles. Un bloc est indexé tout comme le Thread par 3 dimensions dans la grille : blockIdx .x, .y et .z
Première mise en œuvre
Illustrons ce fonctionnement avec la librairie Numba.
Comme nous l’avons vu dans Utiliser des instructions SIMD avec Python, la librairie Numba permet de compiler des fonctions Python et sait tirer parti des jeux d’instructions SIMD quand votre CPU en dispose.
Et elle sait aussi compiler pour GPU avec le decorateur @cuda.jit si vous disposez d’une carte graphique NVIDIA.
Nous allons calculer la fractale de mandelbrot avec un autre exemple de code que nous compilerons grâce à Numba sur CPU (déjà vu dans le TP sur la compilation Just In Time) puis sur GPU.
Dans un premier temps nous allons occulter la question la plus délicate : « Et si mon nombre de cœurs n’est pas multiple de mon nombre de pixels » en fabriquant une image qui respecte ces proportions.
Nous allons aussi ignorer les questions concernant les limites du nombre de blocs dans une grille…
Nous savons que nous disposons de 1024 cœurs au maximum par bloc. Nous créerons des blocs de cette taille.
Commençons par dessiner la fractale puis compilons-là sur CPU.
Reprenons l’exemple de la fractale de Mandelbrot dont le code a été repris de l’article Massively parallel programming with GPUs et que l’on retrouve sur de nombreux autres sites.
import numpy as np
from timeit import default_timer as timer
import matplotlib.pyplot as plt
# color function for point at (x, y)
def mandel(x, y, max_iters):
c = complex(x, y)
z = 0.0j
for i in range(max_iters):
z = z*z + c
if z.real*z.real + z.imag*z.imag >= 4:
return i
return max_iters
def create_fractal(xmin, xmax, ymin, ymax, image, iters):
height, width = image.shape
pixel_size_x = (xmax - xmin)/width
pixel_size_y = (ymax - ymin)/height
for x in range(width):
real = xmin + x*pixel_size_x
for y in range(height):
imag = ymin + y*pixel_size_y
color = mandel(real, imag, iters)
image[y, x] = color
gimage = np.zeros((1024, 1536), dtype=np.uint8)
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
create_fractal(xmin, xmax, ymin, ymax, gimage, iters)
dt = timer() - start
print ("Mandelbrot created on CPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
# https://matplotlib.org/examples/color/colormaps_reference.html
# je vous laisse choisir une belle palette de couleurs, je n'arrive pas à me décider
Mandelbrot created on CPU in 5.265139 s
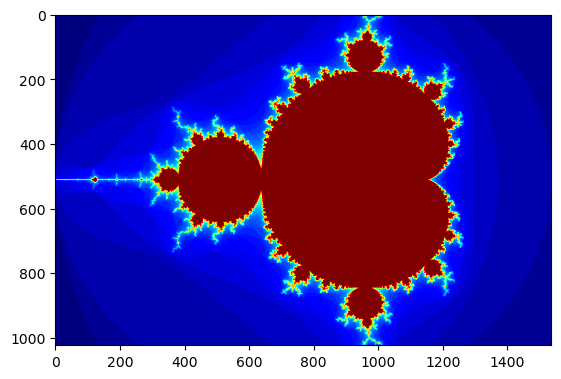
Cinq secondes pour calculer une image de 1024 lignes par 1536 colonnes en pur Python interprété. Ce n’est pas si mal.
Nous pouvons compiler très facilement ce code pour CPU avec numba en ajoutant simplement le décorateur @numba.jit devant chaque fonction.
Celle-ci sera alors transformée pour être compilée lors du premier appel, puis aux appels suivants le code compilé sera utilisé.
Le premier appel est donc toujours plus lent en raison du temps de compilation. L’utilisation de %timeit est plus pertinente pour des mesures de temps : cela dilue le temps de compilation entre tous les appels.
import numba
# color function for point at (x, y)
@numba.jit(nopython=True)
def mandel(x, y, max_iters):
c = complex(x, y)
z = 0.0j
for i in range(max_iters):
z = z*z + c
if z.real*z.real + z.imag*z.imag >= 4:
return i
return max_iters
@numba.jit(nopython=True)
def create_fractal(xmin, xmax, ymin, ymax, image, iters):
height, width = image.shape
pixel_size_x = (xmax - xmin)/width
pixel_size_y = (ymax - ymin)/height
for x in range(width):
real = xmin + x*pixel_size_x
for y in range(height):
imag = ymin + y*pixel_size_y
color = mandel(real, imag, iters)
image[y, x] = color
gimage = np.zeros((1024, 1536), dtype=np.uint8)
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
create_fractal(xmin, xmax, ymin, ymax, gimage, iters)
dt = timer() - start
print ("Mandelbrot created on CPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Mandelbrot created on CPU in 0.333542 s
Relançons une seconde fois le code pour ne plus compter le temps de compilation:
gimage = np.zeros((1024, 1536), dtype=np.uint8)
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
create_fractal(xmin, xmax, ymin, ymax, gimage, iters)
dt = timer() - start
print ("Mandelbrot created on CPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Mandelbrot created on CPU in 0.097677 s
5 / 0.1 # environ 50 fois plus rapide
50.0
Sans mesurer le temps de compilation, le code va 50 fois plus vite.
Mais numba permet de faire encore mieux :
* Les appels à la fonction mandel, qui calcule la couleur d’un pixel, sont indépendants les uns des autres
* Numba peut les paralléliser avec l’option parallel=True transmise au décorateur
* Et l’utilisation de la fonction prange pour Parallel-Range à la place de range qui précise que le contenu de la boucle peut être parallélisé sur différents cœurs.
Nous avons 6 cœurs physiques, nous pourrions encore multiplier la vitesse par 6 !
Exercice
Nous pouvons encore gagner plus en demandant à Numba de paralléliser les appels à mandel avec l’option @numba.jit(parallel=True)
Essayez de le faire.
# A vous de jouer !
Solution
import numba # color function for point at (x, y) @numba.jit(nopython=True) def mandel(x, y, max_iters): c = complex(x, y) z = 0.0j for i in range(max_iters): z = z*z + c if z.real*z.real + z.imag*z.imag >= 4: return i return max_iters @numba.jit(parallel=True, nopython=True) def create_fractal(xmin, xmax, ymin, ymax, image, iters): height, width = image.shape pixel_size_x = (xmax - xmin)/width pixel_size_y = (ymax - ymin)/height for x in numba.prange(width): real = xmin + x*pixel_size_x for y in numba.prange(height): imag = ymin + y*pixel_size_y color = mandel(real, imag, iters) image[y, x] = color
gimage = np.zeros((1024, 1536), dtype=np.uint8)
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
create_fractal(xmin, xmax, ymin, ymax, gimage, iters)
dt = timer() - start
print ("Mandelbrot created on CPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Mandelbrot created on CPU in 0.375268 s
Et une seconde fois, pour ne pas compter le temps de compilation :
gimage = np.zeros((1024, 1536), dtype=np.uint8)
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
create_fractal(xmin, xmax, ymin, ymax, gimage, iters)
dt = timer() - start
print ("Mandelbrot created on CPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Mandelbrot created on CPU in 0.017792 s
5 / 0.017
294.11764705882354
Nous frôlons le ratio théorique de 50 * 6 = 300 fois plus rapide que la version originale en pur Python !
Vraiment impressionnant quand la pratique du matériel respecte la théorie !
N’est-ce pas ?
Nous allons maintenant compiler ces fonctions avec Numba pour CUDA/NVIDIA.
Avant d’utiliser numba+cuda, nous devons libérer le contexte d’exécution – notion qui sera abordée avec le TP PyOpenCL/PyCUDA – pris par PyCuda sur le GPU car il va gêner le fonctionnement de numba avec une erreur de type Non Primary Context
ctx = drv.Context.get_current()
drv.Context.detach(ctx)
Nous pouvons maintenant continuer.
La fonction mandel est appelée pour chaque pixel de l’image. Ce sera notre Thread qui sera exécuté sur chacun des cœurs.
Il suffit de la préfixer avec le décorateur @cuda.jit et de lui passer le paramètre device=True qui signifie qu’elle sera utilisée comme fonction device, c’est-à-dire s’exécutant sur le GPU.
from numba import cuda
import numpy as np
from timeit import default_timer as timer
@cuda.jit(device=True)
def mandel(x, y, max_iters):
c = complex(x, y)
z = 0.0j
for i in range(max_iters):
z = z*z + c
if z.real*z.real + z.imag*z.imag >= 4:
return i
return max_iters
Voici qui est fait pour la première. Ce n’est pas plus compliqué que pour le CPU.
La seconde fonction est la fonction qui lance la fonction device. C’est notre fonction kernel qui sera exécutée sur l’hôte.
La version compilée sera exécutée pour chaque Thread de la grille CUDA.
Une fois décorée, son invocation est particulière car elle va être exécutée pour chacun des Threads :
<fonction kernel>[grid_dim, block_dim](<params>)
grid_dimest un tuple indiquant le nombre de blocs par grille en colonnes et lignes (et profondeur si besoin).
ATTENTION CUDA utilise les colonnes en première dimension, contrairement à NumPy qui utilise les lignes.bloc_dimest un tuple indiquant le nombre de Threads par bloc
Il convient donc de définir la structure de notre grille et de nos blocs.
Nous avons choisi de travailler avec une image dont les dimensions sont compatibles avec la taille maximale des blocs dans ce premier exemple, pour plus de simplicité.
Nous savons que nous ne pouvons pas utiliser plus de 1024 Threads par bloc. Ceci permet d’avoir des blocs carrés de 32×32, mais nous aurions pu chosir 64×16 ou tout autre couple dont le produit est 1024.
Si nous restons sur des blocs carrés (32×32), sachant que l’image fait 1536 colonnes et 1024 lignes, nous pouvons alors définir une grille de 48 blocs en colonnes et 32 lignes.
1536 / 32, 1024 / 32
(48.0, 32.0)
Maintenant, il convient de restructurer le code de la fonction Kernel.
Celle-ci, auparavant, utilisait 2 boucles imbriquées pour parcourir toutes les colonnes puis les lignes de l’image et appeler la fonction mandel pour chaque pixel ainsi parcouru.
Avec CUDA, elle sera appelée pour chaque Thread de chaque bloc de la grille. Ces boucles ne sont plus nécessaires et doivent disparaître.
Nous devons donc transformer le code pour calculer les coordonnées du pixel en fonction des coordonnées du thread dans le bloc et du bloc dans la grille.
Les variables
cuda.threadIdx.xetcuda.threadIdx.ycontiennent les coordonnées du Thread dans le bloc.
De 0 à 31 dans notre casLes variables
cuda.blockIdx.xetcuda.blockIdx.ycontiennent les coordonnées du bloc dans la grille.
De 0 à 47 dans notre cas pour les colonnes et de 0 à 31 pour les lignes.Les variables
cuda.blockDim.xetcuda.blockDim.ycontiennent les dimensions du bloc. Dans notre cas : 32 colonnes et 32 lignes
Avec ces variables, nous pouvons donc reconstituer les coordonnées du pixel sur lequel la fonction device est exécutée.
# Son code peut être transformé ainsi:
@cuda.jit()
def create_fractal(xmin, xmax, ymin, ymax, image, iters):
height, width = image.shape
pixel_size_x = (xmax - xmin)/width
pixel_size_y = (ymax - ymin)/height
tx = cuda.threadIdx.x # abscisse de 0 à 31 du Thread dans le block
ty = cuda.threadIdx.y # idem en ligne pour l'ordonnée
bx = cuda.blockIdx.x # abscisse de 0 à 47 du bloc dans la grille
by = cuda.blockIdx.y # ordonnée de 0 à 31 du bloc dans la grille
# Block width, i.e. number of threads per block
bw = cuda.blockDim.x # largeur du bloc : nombre de Threads en colonnes dans le bloc
bh = cuda.blockDim.y # longueur du bloc : nombre de Threads en lignes dans le bloc
# Recalculons les valeurs de x, y, imag et real avec les coordonnées des Threads/Blocs
"""
# Que fait le code original ?
for x in range(width): # Il parcourt les colonnes de l'image avec x
real = xmin + x*pixel_size_x # Puis il calcule real en fonction de l'abscisse x dans l'image
for y in range(height): # Pour chaque colonne il parcourt les lignes
imag = ymin + y*pixel_size_y # Puis il calcule imag en fonction de l'ordonnée y dans l'image
color = mandel(real, imag, iters) # et appelle mandel pour chaque pixel avec real et imag
image[y, x] = color
"""
# Maintenant, cette fonction sera exécutée sur un cœur, via un Thread, pour chaque Thread de chaque bloc de la grille
# La position du cœur/thread dans son bloc et du bloc dans la grille indiqueront les coordonnées du pixel calculé
# L'abscisse devient : position en X du Thread dans le bloc + position du bloc en X dans la grille * largeur du bloc en colonne
# Par exemple, le 36ème pixel (abscisse x=35) aura pour abscisse de thread 3 dans le bloc 1 dont la largeur est 32 : 3 + 1 * 32 = 35
x = tx + bx * bw
y = ty + by * bh
real = xmin + x*pixel_size_x
imag = ymin + y*pixel_size_y
image[y, x] = mandel(real, imag, iters)
Ici le code est vraiment très différent, c’est une fonction elementwize qui s’applique élément par élément.
Pour compiler votre code Python avec Numba sur GPU, il faut vraiment passer par cette étape de restructuration/atomisation du code par élément du tableau.
Ceci le rend moins portable, mais pas plus long.
L’effort intellectuel est conséquent, prenez le temps de dessiner le processus dans le code si vous avez du mal à le comprendre.
Maintenant, exécutons-le pour vérifier que cela fonctionne bien et mesurer s’il mérite la peine que nous nous sommes donnés…
import matplotlib.pyplot as plt
gimage = np.zeros((1024, 1536), dtype=np.uint8)
blockdim = (32, 32)
griddim = (48, 32) # la grille utilise les colonnes en 1ere coordonnée
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
d_image = cuda.to_device(gimage)
create_fractal[griddim, blockdim](xmin, xmax, ymin, ymax, d_image, iters)
gimage = d_image.copy_to_host()
dt = timer() - start
print("Mandelbrot created on GPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Mandelbrot created on GPU in 0.219687 s
N’oubliez pas la seconde exécution pour ne pas compter le temps de compilation…
import matplotlib.pyplot as plt
gimage = np.zeros((1024, 1536), dtype=np.uint8)
blockdim = (32, 32)
griddim = (48, 32) # la grille utilise les colonnes en 1ere coordonnée
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
d_image = cuda.to_device(gimage)
create_fractal[griddim, blockdim](xmin, xmax, ymin, ymax, d_image, iters)
gimage = d_image.copy_to_host()
dt = timer() - start
print("Mandelbrot created on GPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Mandelbrot created on GPU in 0.006086 s
Nous sommes près de 600 fois plus rapides que le code Python d’origine, et cela inclut le temps de transfert CPU-GPU qui est coûteux.
5 / 0.006
833.3333333333334
Si vous souhaitez juste mesurer le temps de la fonction create_fractal seule il faut savoir que son appel n’est pas bloquant. Pour votre CPU le GPU est un co-processeur, un périphérique, il lui transmet les ordres et continue sa vie sans attendre le retour.
Donc %timeit create_fractal[griddim, blockdim](xmin, xmax, ymin, ymax, d_image, iters) seul n’indiquerait que le temps de lancement de la fonction, même si elle prend des heures à s’exécuter.
Il faut appeler la fonction cuda.synchronize() pour demander au CPU d’attendre la fin des tâches du GPU.
%%timeit
create_fractal[griddim, blockdim](xmin, xmax, ymin, ymax, d_image, iters)
numba.cuda.synchronize()
2.72 ms ± 20.3 μs per loop (mean ± std. dev. of 7 runs, 100 loops each)
5 / 0.0027 # 1800 fois plus rapide ! Merci numba.
1851.8518518518517
Si après le calcul vous transférez des données du GPU au CPU cela bloque aussi votre CPU sur l’attente du résultat et les temps affichés sont alors fiables.
Nous avons maintenant un code plus de 1700 fois plus rapide que celui initialement réalisé en pur python. Cela en valait la peine !
Magnifique !
Mais, maintenant, comment dimensionner la grille si le tableau n’est pas multiple du nombre de cœurs ?
D’autant plus que dans notre exemple nous utilisons des blocs de 1024 cœurs, or nous avons 2304 cœurs, ce qui signifie que nous ne pouvons faire que 2 blocs et que potentiellement 280 cœurs ne sont pas utilisés.
Notre découpage n’est pas idéal.
Bien utiliser les Streaming Multiprocessors (SM)
Le code fonctionne bien mais n’est pas encore parfait : Nous ne gérons pas correctement les processeurs de flux (Streaming Multiprocessors).
Le code exécuté sur la carte GPU l’est au travers des SM qui ne peuvent utiliser qu’un nombre maximal (puissance de 2 – à vérifier) de Threads. Le nombre de SM est aussi limité.
D’autre part, ici tout s’est bien passé car la taille de l’image est multiple du nombre de threads par bloc. Mais comment gérer notre problème si l’image possède une ligne et une colonne de plus ?
L’article Writing cuda kernels de la présentation GTC Numba l’explique.
Pour comprendre l’algorithme à mettre en œuvre dans ce cas, nous allons imaginer cette configuration très simple :
- Une carte graphique :
- Dotée de 6 cœurs uniquement
- Pouvant totaliser des blocs d’au plus deux Threads par sm
- Ne pouvant dépasser 3 SM
- Une matrice de 11 colonnes et 2 lignes sur laquelle nous souhaitons appliquer un calcul elementwise.
L’idée va être la suivante :
* On crée a minima autant de blocs qu’il y a de SM, donc 3 ici.
Que nous nommons ici B0, B1 et B2, ils représentent la grille !
* On choisit une disposition des threads dans un bloc qui semble cohérente avec la matrice, dans notre schéma ci-dessous : 2 colonnes, 1 ligne.
Avec 3 blocs de 2 Threads, cela fait 6 Threads dans la grille pour 22 éléments :
- Le Bloc B0 (en vert) possède 2 Threads numérotés t0, t1 dans celui-ci
- Le Bloc B1 (en jaune) possède 2 Threads numérotés t0, t1 dans celui-ci
- Le Bloc B2 (en bleu) possède 2 Threads numérotés t0, t1 dans celui-ci
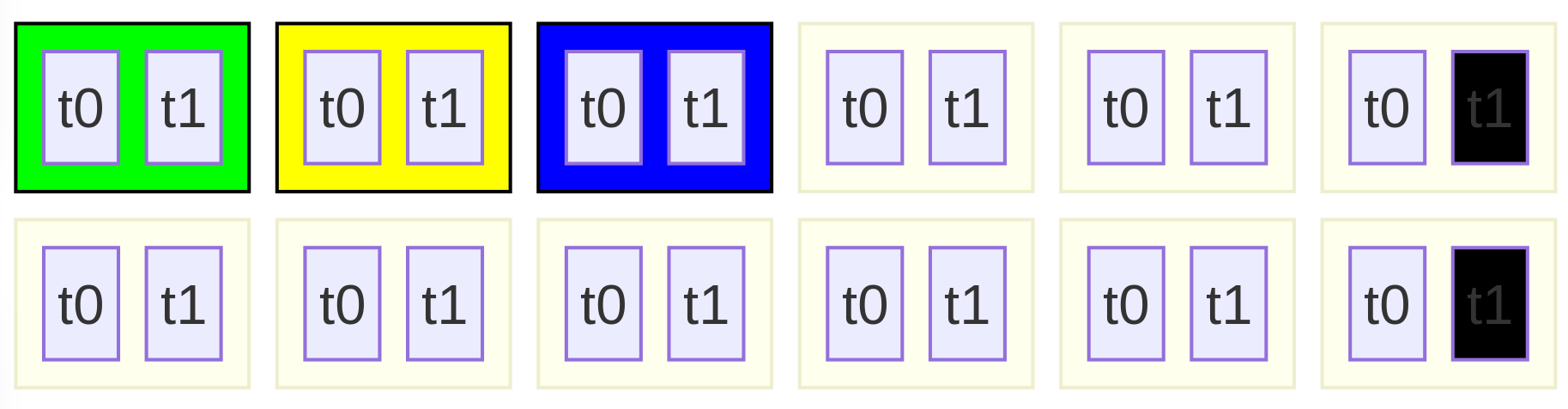
3 blocs de 2 threads
La grille est plus petite que la matrice à calculer !
Beaucoup d’éléments passeraient à la trappe si nous nous arrêtions là…
Aussi chaque Thread ne s’occupera pas dans son bloc que d’un seul élément du tableau mais de plusieurs.
Dans un bloc, chaque Thread s’occupera :
* De l’élément qu’il désigne
* Le Thread t0 du bloc B0 s’occupera de l’élément en 0,0
* Le Thread t1 du bloc B0 s’occupera de l’élément en 1,0 # colonne d’abord avec CUDA
* Le Thread t0 du bloc B1 s’occupera de l’élément en 2,0
* Le Thread t1 du bloc B1 s’occupera de l’élément en 3,0
* Le Thread t0 du bloc B2 s’occupera de l’élément en 4,0
* Le Thread t1 du bloc B2 s’occupera de l’élément en 5,0
* Mais aussi des éléments situés à la même position décalés de la hauteur et largeur de la grille en éléments (6, 1) tant que les dimensions de la matrice ne sont pas dépassées
* Le Thread t0 du bloc B0 s’occupera donc aussi des éléments (0, 1), (6, 0) et (6, 1)
* Le Thread t1 du bloc B0 s’occupera donc aussi des éléments (1, 1), (7, 0) et (7, 1)
* …
* Le Thread t1 du bloc B2 s’occupera donc aussi de l’élément (5, 1) et ce sera tout car après il serait hors de la matrice
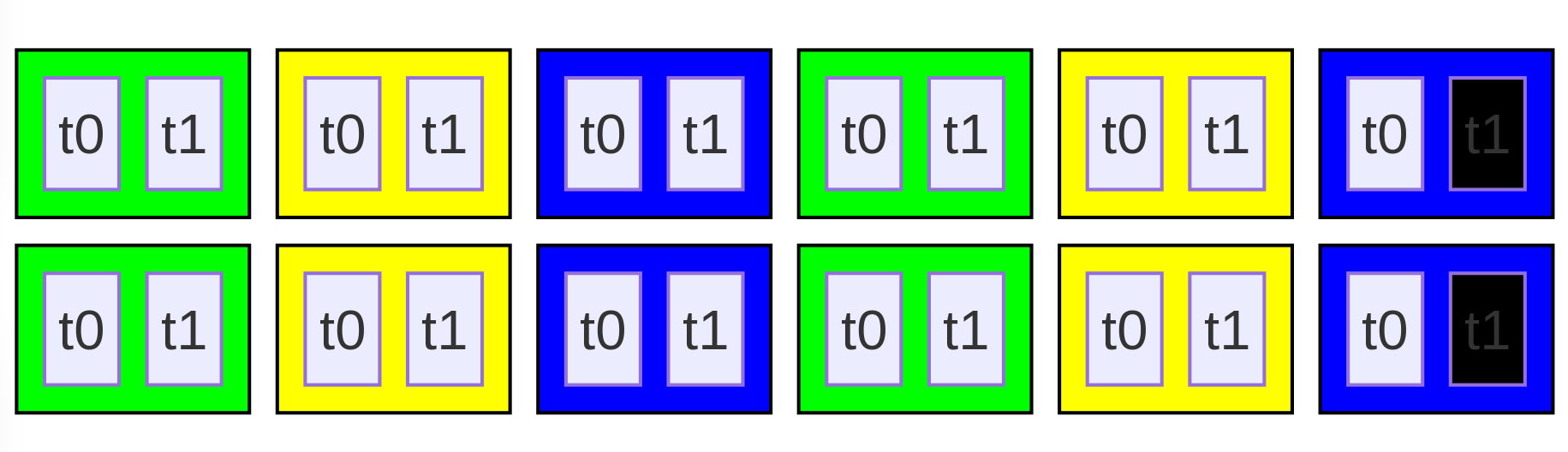
3 blocs de 2 threads :
Voici l’exemple de writing cuda kernels qui additionne deux vecteurs de 100 000 éléments, nombre d’éléments qui n’est pas un multiple du nombre de cœurs de la carte…
from numba import cuda
@cuda.jit
def add_kernel(x, y, out):
tx = cuda.threadIdx.x # this is the unique thread ID within a 1D block
bx = cuda.blockIdx.x # Similarly, this is the unique block ID within the 1D grid
block_width = cuda.blockDim.x # number of threads per block
grid_width = cuda.gridDim.x # number of blocks in the grid
start = tx + bx * block_width
step_x = block_width * grid_width
# assuming x and y inputs are same length
for i in range(start, x.shape[0], step_x):
out[i] = x[i] + y[i]
import cupy as cp
n = 100_000
x = cp.arange(n).astype(np.float32)
y = 2 * x
out = cp.empty_like(x)
threads_per_block = 64
blocks_per_grid = 36 # Utilisation de 30 SM
add_kernel[blocks_per_grid, threads_per_block](x, y, out)
print(out[:10])
print(out[-10:])
[ 0. 3. 6. 9. 12. 15. 18. 21. 24. 27.]
[299970. 299973. 299976. 299979. 299982. 299985. 299988. 299991. 299994.
299997.]
Ici, le nombre de blocs est égal au nombre de SM : 36.
Il y a 64 Threads par SM, soit 36*64 = 2304 cœurs de la carte graphique utilisés
L’idée est qu’un Thread ne mettra pas à jour qu’une seule case mémoire, mais sa case mémoire et toutes les suivantes décalées du nombre total de cœurs utilisés (step_x)
Comme la boucle for itère de la position courante du thread jusqu’à la fin du tableau avec un pas de step_x nous ne risquons pas de dépasser la fin du tableau.
Voici les recommandations des développeurs Numba/CUDA :
- La taille d’un bloc doit être un multiple de 32 Threads, avec des tailles de bloc typiques entre 128 et 512 Threads par Bloc.
- La taille de la grille doit permettre d’utiliser l’intégralité du GPU dans la mesure du possible
N’hésitez pas à avoir plus de blocs que de SM : 2 à 4 fois plus est un bon point de départ - L’exécution répétée de la fonction Kernel pour démarrer chaque Thread génère un surcout proportionnel au nombre de Blocs/Threads.
Il est donc préférable de ne pas lancer une grille dont le nombre de Threads est égal au nombre d’éléments d’entrée lorsque la taille de l’entrée est très importante.
*Dans notre premier exemple avec la fractale, c’est exactement ce que nous avons fait et ce qu’il ne fallait pas faire !
Exercice
Reprenez le code de la fonction kernel pour l’adapter à ces recommandations et permettre de calculer sur une image dont les dimensions ne sont pas des multiples du nombre de cœurs.
# A vous de jouer !
Solution
# Son code peut être transformé ainsi: @cuda.jit() def create_fractal(xmin, xmax, ymin, ymax, image, iters): height, width = image.shape pixel_size_x = (xmax - xmin)/width pixel_size_y = (ymax - ymin)/height tx = cuda.threadIdx.x # abscisse de 0 à 31 du thread dans le block ty = cuda.threadIdx.y # idem en ligne pour l'ordonnée bx = cuda.blockIdx.x # abscisse de 0 à 47 du bloc dans la grille by = cuda.blockIdx.y # ordonnée de 0 à 31 du bloc dans la grille # Block width, i.e. number of threads per block bw = cuda.blockDim.x # largeur du bloc : nombre de threads en colonnes dans le bloc bh = cuda.blockDim.y # longueur du bloc : nombre de threads en lignes dans le bloc gw = cuda.gridDim.x gh = cuda.gridDim.y step_x = bw * gw step_y = bh * gh start_x = x = tx + bx * bw start_y = ty + by * bh for x in range(start_x, width, step_x): # Il parcourt les colonnes de l'image avec x real = xmin + x*pixel_size_x # Puis il calcule real en fonction de l'abscisse x dans l'image for y in range(start_y, height, step_y): # Pour chaque colonne il parcourt les lignes imag = ymin + y*pixel_size_y # Puis il calcule imag en fonction de l'ordonnée y dans l'image color = mandel(real, imag, iters) # et appelle mandel pour chaque pixel avec real et imag image[y, x] = color
import matplotlib.pyplot as plt
gimage = np.zeros((1024+1, 1536+1), dtype=np.uint8)
blockdim = (8, 8)
griddim = (72, 4) # la grille utilise les colonnes en 1ere coordonnée
xmin, xmax, ymin, ymax = np.array([-2.0, 1.0, -1.0, 1.0]).astype('float32')
iters = 50
start = timer()
d_image = cuda.to_device(gimage)
create_fractal[griddim, blockdim](xmin, xmax, ymin, ymax, d_image, iters)
gimage = d_image.copy_to_host()
dt = timer() - start
print("Mandelbrot created on GPU in %f s" % dt)
plt.imshow(gimage, cmap='jet');
Bienvenue au pays des développeurs Python sur GPU !
Pour aller plus loin
Il existe de nombreux tutoriaux et livres sur le calcul pour GPU avec Python.
Les articles du compte GitHub de Jean François Puget constituent d’excellentes lectures :
Deux livres intéressants sur les GPU :
Hands-On GPU Computing with Python

Source PacktPub Auteur : Avimanyu Bandyopadhyay
En quelques mots :
- Il prend le temps de présenter l’état de l’art de la discipline et les applications industrielles
- Il vous aide à choisir une solution matérielle
- Il présente bien les architectures NVIDIA et AMD
- De nombreuses librairies sont abordées
- Enfin, il a été publié en mai 2019, il reste à jour
- Il est un peu juste si vous souhaitez creusez le sujet de la programmation GPU
Hands-On GPU Programming with Python and CUDA

Source PacktPub Auteur : Tuomanen
En quelques mots :
- Il est spécifique à NVIDIA mais va bien plus loin dans la programmation CUDA
- Il reste majoritairement focalisé sur PyCUDA, donc il y aura aussi du code en C : mais c’est ce qu’il annonce
- Il propose pas mal d’exercices pratiques
Licence
Ce tutoriel et les images qu’il a générées sont sous licence : CC BY-NC-SA 4.0
Vous pouvez copier, modifier et distribuer ce tutoriel sous réserve des conditions de la licence, dont :
- Respecter le crédit de l’auteur : Gaël Pegliasco
- Respecter le crédit des images externes au cours ayant permis de l’illustrer qui possèdent leur propre licence
- Ne pas l’utiliser dans un but commercial
- Partager vos adapatations sous la même licence
Il est accessible sous forme de Notebook Jupyter sous GitHub à cette url : https://github.com/MordicusEtCubitus/CoursPython/tree/master/gpu
En savoir plus
Formations associées
Ateliers Data Science
Parcours complet Data Science
Nantes - Toulouse - Paris ou distanciel Nous contacter
Voir la Parcours complet Data ScienceFormations IA / Data Science
Formation Python Calcul Scientifique
Toulouse Du 3 au 5 février 2026
Voir la Formation Python Calcul ScientifiqueFormations IA / Data Science
Formation Python scientifique
Aucune session de formation n'est prévue pour le moment.
Pour plus d'informations, n'hésitez pas à nous contacter.
Voir la Formation Python scientifiqueActualités en lien
Calculez sur GPU avec Python – Partie 2/3
Data Science
11/02/2025

Calculez sur GPU avec Python – Partie 1/3
Data Science
04/02/2025

Notre expertise en Data science
Data Science
27/03/2019
L'IA est un domaine vaste et nos différents projets nous ont permis de monter en compétences sur plusieurs sujets. Cet article vous présente les thématiques pour lesquelles nous pouvons vous offrir notre expertise.